Gli scienziati stanno trovando il modo di rendere questi nanodispositivi programmabili stabili e funzionali all'interno delle cellule viventi. In caso di successo, potrebbero rivoluzionare il modo in cui interfacciamo e controlliamo la biologia a livello molecolare
I circuiti di dislocamento del filo di DNA si avvicinano a diventare macchine cellulari.
Una recente recensione pubblicata su Intelligent Computing (1), descrive i principali progressi nello sforzo di portare i circuiti di calcolo del DNA nelle cellule viventi. Gli autori descrivono come i nano dispositivi dinamici alimentati dalle reazioni di spostamento del filamento di DNA possano presto eseguire il calcolo, il rilevamento e il controllo in tempo reale all'interno dei sistemi biologici, aprendo le porte a una nuova generazione di “robot molecolari” che interagiscono direttamente con gli ambienti cellulari.
Come lo spostamento del filo del DNA alimenta la logica molecolare
Al centro di questa tecnologia ci sono i circuiti di spostamento del filamento di DNA, un componente chiave della nanotecnologia dinamica del DNA. Questi circuiti usano lo spostamento del filamento mediato da un tocco: un processo in cui un filamento di DNA in arrivo si lega a una breve regione esposta chiamata toehold, e quindi sposta un filo esistente attraverso la migrazione della sezione.
I sistemi di base come porte seesaw e reazioni della catena di ibridazione consentono operazioni logiche complesse e amplificazione del segnale, mentre le porte cooperative richiedono più input per produrre un output, consentendo un controllo sofisticato. Questi singoli componenti possono essere combinati in reti più grandi che simulano percorsi di reazione chimica formale. Lo spostamento del filo di DNA si collega anche a nanodispositivi strutturali come origami di DNA e assemblaggi di DNA, consentendo cambiamenti di forma controllata e espandendo la gamma di applicazioni biologiche.
Input biologici per il rilevamento basato sul DNA
Secondo gli autori, «Le reazioni di spostamento del filo di DNA possono essere innescate da componenti biologici come acidi nucleici, piccole molecole, proteine e ioni».
Gli acidi nucleici, come il DNA e l'RNA, servono come input diretti sfruttando progetti di substrato complementari, consentendo applicazioni nell'analisi del trascrittoma e nel monitoraggio delle cellule vive. Il rilevamento degli input può essere ottenuto attraverso aptameri, che sono sequenze di acido nucleico a singolo filamento che si legano a bersagli o ligandi con alta affinità e specificità.
Per collegare gli aptameri ai moduli di spostamento del filamento di DNA, vari metodi come gli aptameri di commutazione di struttura, appigli associativi, appigli nascosti, appigli remoti, appigli transitori, legatura chimica, metallo-toeholds e DNAzymes sono stati sviluppati per garantire il segnale preciso: transduzioni da bersagli biologici a circuiti a valle.
Sfide e soluzioni per la funzionalità in cellula
Attualmente, lo spostamento del filo del DNA viene applicato principalmente in vitro e la sua applicazione in vivo affronta importanti sfide, tra cui una rapida degradazione da parte degli enzimi degradanti del DNA. Per migliorare la stabilità, i ricercatori hanno esplorato modifiche strutturali come protezioni terminali come forcine e siti di legame proteico, nonché modifiche chimiche come 2’-O-metilazione.
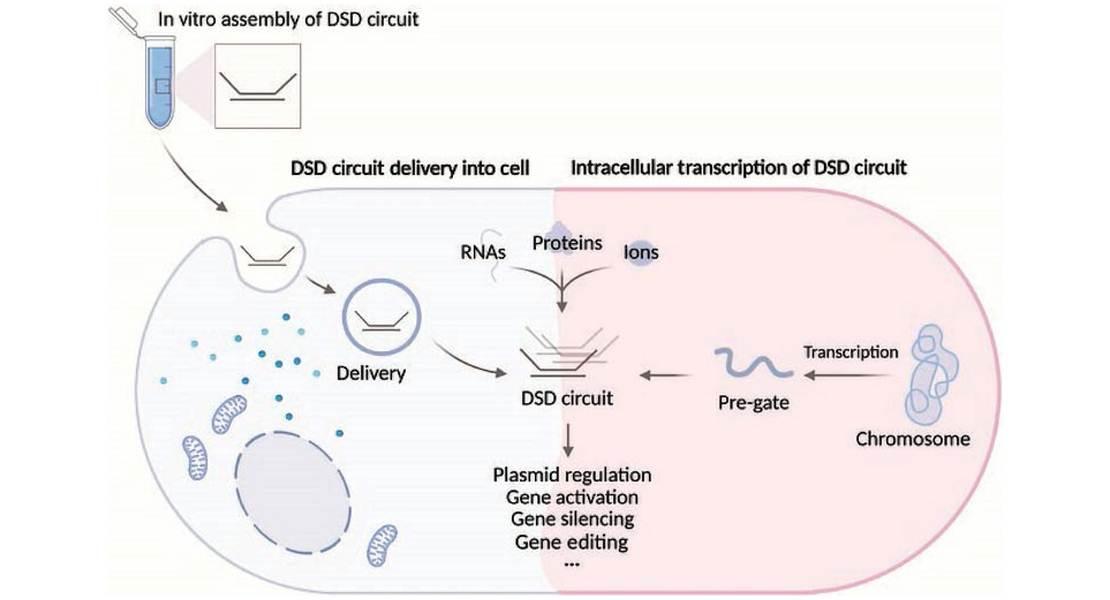
Poiché la maggior parte delle cellule respinge naturalmente il DNA, la consegna di questi nano dispositivi in cellule richiede tecniche specializzate, come metodi di trasfezione e protocolli di trasformazione. Una volta all'interno, fattori cellulari come la concentrazione di sale, l'affollamento molecolare e gli ambienti eterogenei influenzano le reazioni di spostamento del filo. Per superare i limiti della consegna diretta, i ricercatori stanno anche sviluppando nanodispositivi di RNA trascrivibili codificati in plasmidi o cromosomi, consentendo alle cellule di esprimere questi circuiti.
Verso macchine e biocomputer intelligenti di DNA
Lo spostamento del filo di DNA è stato applicato all'innovazione dei modelli computazionali. Integrando i principi computazionali con spostamento del filo di DNA, gli algoritmi strutturati del calcolo tradizionale possono essere combinati con processi biochimici casuali e reazioni chimiche nei sistemi biologici, consentendo modelli di calcolo biocompatibili. In futuro, lo spostamento del filo di DNA può consentire alle nanomachine del DNA che agiscono autonomamente di manipolare con precisione i processi biologici, portando a salti quantistici nella ricerca sulla salute e nelle scienze della vita.
Riferimenti:
(1) From the Test Tube to the Cell: A Homecoming for DNA Computing Circuits?
Descrizione foto: Sul lato sinistro, i circuiti di DNA preassemblati in vitro vengono consegnati nelle cellule viventi. In alternativa, sul lato destro, i pregate di RNA vengono trascritti autonomamente dal cromosoma o dai plasmidi per essere quindi convertiti in circuiti di RNA funzionali. In entrambi i casi, i circuiti funzionali rilevano quindi lo stato trascrizionale e metabolico della cellula ed eseguono l'integrazione del segnale e altri calcoli per attuare in definitiva diversi processi biologici come la regolazione genica post -trascrizionale. - Credit: Hyeyun Jung et al.
Autore traduzione riassuntiva e adattamento linguistico: Edoardo Capuano / Articolo originale: DNA Circuits Come Alive: Scientists Build Molecular Robots Inside Living Cells